Roche uPath PD-L1 image analysis for NSCLC IVD Algorithm Instrukcja obsługi
- Typ
- Instrukcja obsługi

Algorytm uPath PD-L1
(SP263) do analizy obrazów
niedrobnokomórkowego raka płuc —
podręcznik do algorytmu


Spis treści
Wstęp 1
Podsumowanie i objaśnienie działania algorytmu 2
Przeznaczenie 3
Przeznaczenie produktu 3
Przeznaczenie podręcznika do algorytmu 3
Znaczenie kliniczne 4
Zasady testu 5
Ograniczenia metody 6
Bezpieczeństwo danych 7
Przebieg pracy z wykorzystaniem algorytmu uPath PD-L1 do analizy
obrazów raka NSCLC 8
Przebieg pracy wykonywanej przez patomorfologa 9
Analiza obrazu pod kątem białka PD-L1 12
Ocena pod kątem białka PD-L1 15
Ocena wybarwienia za pomocą algorytmu PD-L1 15
Charakterystyka barwienia 15
Charakterystyka wyników 26
Porównanie metod 26
Badania odtwarzalności wyników uzyskiwanych przez patomorfologów 27
Badania odtwarzalności wyników uzyskiwanych przy użyciu skanera 28
Rozwiązywanie problemów 29
Piśmiennictwo 33


Wstęp
Oprogramowanie Roche uPath enterprise software
(oprogramowanie uPath enterprise software) wyposażone
w algorytm uPath PD-L1 (SP263) do analizy obrazów
niedrobnokomórkowego raka płuc (NSCLC) (algorytm uPath PD-L1
(SP263) do analizy obrazów raka NSCLC) to oprogramowanie,
które zaprojektowano w celu wspomagania ilościowej oceny
ekspresji białka w histologicznych skrawkach barwionych metodą
immunohistochemiczną (IHC) uzyskanych z prawidłowych i
nowotworowych tkanek utrwalonych w formalinie i zatopionych
w parafinie (FFPE).
Oprogramowanie uPath enterprise software jest całościowym
cyfrowym narzędziem programowym do zastosowania w
patologii, które umożliwia laboratoriom patologicznym pobieranie,
przeglądanie, analizowanie i przekazywanie cyfrowych obrazów
próbek patologicznych, zarządzanie nimi i tworzenie raportów
na ich podstawie. Za pomocą oprogramowania uPath enterprise
software patomorfolog może przeglądać obrazy cyfrowe w
różnych powiększeniach, dodawać adnotacje, dokonywać
pomiarów na skrawkach tkankowych, przeprowadzać analizę
obrazową i generować raporty.
Uwaga: algorytm uPath PD-L1 (SP263) do analizy obrazów raka
NSCLC to uzupełniająca, wspomagana komputerowo metoda
przeznaczona do ułatwiania procesu rejestracji obrazów z
podstawowych szkiełek mikroskopowych, na których znajdują się
próbki tkanek barwione metodą IHC, oraz wykonywania pomiarów
na tych obrazach w celu określenia obecności białka PD-L1.
Patomorfolog jest odpowiedzialny za weryfikację zgodności
wyników przy użyciu odpowiednich kontroli zgodnie z opisem
w arkuszu metody dla przeciwciała VENTANA PD-L1 (SP263)
Rabbit Monoclonal Primary Antibody (dostępny pod adresem
www.ventana.com), aby zagwarantować ważność wyników analiz
obrazowych.
Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu 1

2 Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu
Podsumowanie i objaśnienie działania
algorytmu
W przypadku zastosowań do analizy obrazowej patomorfolog
może używać oprogramowania uPath enterprise software
do wybierania i obrysowywania jednego lub kilku obszarów
zainteresowania (ROI), przy czym każdy obszar ROI może być
wyświetlany w różnych powiększeniach, a następnie analizowany
przez algorytm uPath PD-L1 (SP263) do analizy obrazów raka
NSCLC. Generowana jest łączna liczba docelowych komórek
nowotworowych (TC), a następnie komórki TC są przydzielane
do grupy komórek wybarwionych lub niewybarwionych.
Algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC
dzieli wybarwione komórki TC przez łączną liczbę komórek TC
(wybarwionych i niewybarwionych) w celu wygenerowania
wyniku określającego odsetek komórek TC dodatnich względem
białka PD-L1 w skali 0–100%. Algorytm uPath PD-L1 (SP263)
do analizy obrazów raka NSCLC może wygenerować wynik dla
określonego obszaru ROI lub sumaryczny wynik dla wszystkich
obszarów ROI zaznaczonych na danym preparacie. Chociaż
algorytm wykrywa komórki, które nie są zmienione nowotworowo,
w ramach ogólnej analizy, nakładka i dane wyjściowe
wskazują jedynie komórki TC użyte do obliczenia wyniku
określającego odsetek dodatnich komórek TC. Patomorfolog
może zaakceptować wynik podany przez algorytm uPath PD-L1
(SP263) do analizy obrazów raka NSCLC lub może zastąpić ten
wynik innym wynikiem. Patomorfolog musi dokonać dokładnego
przeglądu komórek TC określonych przez algorytm jako komórki
dodatnie lub ujemne i potwierdzić, że algorytm zadziałał
prawidłowo, lub ręcznie wprowadzić wynik. Algorytm uPath
PD-L1 (SP263) do analizy obrazów raka NSCLC nie dokonuje
niezależnej interpretacji danych i z tego względu powinien być
używany wyłącznie przez wykwalifikowanych patomorfologów, w
połączeniu z wynikami badania histologicznego, odpowiednimi
informacjami klinicznymi oraz odpowiednimi kontrolami. Algorytm
ten został zaprojektowany jako pomoc dla patomorfologów w
ocenie ekspresji białka PD-L1 na poziomie 50%.

Przeznaczenie
Przeznaczenie podręcznika do algorytmu
Dokument Algorytm uPath PD-L1 (SP263) do analizy obrazów
raka NSCLC — podręcznik do algorytmu (podręcznik do algorytmu)
opracowano w celu:
• udostępnienia ogólnych informacji dotyczących przeznaczenia
algorytmu uPath PD-L1 (SP263) do analizy obrazów raka
NSCLC, zasad oraz ograniczeń metody;
• zdefiniowania wymaganych materiałów, a także wymogów w
zakresie technologii informacyjnej, bezpieczeństwa danych
oraz sieci;
• przedstawienia kroków obsługi algorytmu uPath PD-L1 (SP263)
do analizy obrazów raka NSCLC;
• udostępnienia obrazów fotograficznych ilustrujących sposób
korzystania z algorytmu uPath PD-L1 (SP263) do analizy
obrazów raka NSCLC;
• udostępnienia patomorfologom narzędzia ułatwiającego
korzystanie z algorytmu uPath PD-L1 (SP263) do analizy
obrazów raka NSCLC na skrawkach FFPE tkanki płuca z
rakiem NSCLC barwionych przy użyciu testu VENTANA PD-L1
(SP263) Assay;
• udostępnienia przykładowych obrazów trudnych przypadków
w celu przedstawienia sposobu korzystania z algorytmu uPath
PD-L1 (SP263) do analizy obrazów raka NSCLC do oceny
takich przypadków;
• przedstawienia charakterystyki wyników algorytmu uPath
PD-L1 (SP263) do analizy obrazów raka NSCLC.
Przeznaczenie produktu
Algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC
jest przeznaczony do wspomagania patomorfologów w detekcji
i pomiarze półilościowym białka PD-L1 w utrwalonych w
formalinie i zatopionych w parafinie skrawkach tkanki płuca z
rakiem NSCLC. W przypadku stosowania algorytmu wraz z testem
VENTANA PD-L1 (SP263) Assay algorytm służy do pomocy w
identyfikacji pacjentów z rakiem NSCLC do leczenia za pomocą
terapii stosowanych w przypadku ekspresji białka PD-L1 w ≥50%
komórek TC (punkt odcięcia) zgodnie z zatwierdzoną ChPL.
Uwaga: algorytm uPath PD-L1 (SP263) do analizy obrazów raka
NSCLC to uzupełniająca, wspomagana komputerowo metoda
przeznaczona do ułatwiania procesu rejestracji obrazów z
podstawowych szkiełek mikroskopowych, na których znajdują się
próbki tkanek barwione metodą IHC, oraz wykonywania pomiarów
na tych obrazach w celu określenia obecności białka PD-L1.
Patomorfolog jest odpowiedzialny za weryfikację zgodności
wyników i stosowanie odpowiednich kontroli zgodnie z opisem
w arkuszu metody dla testu VENTANA PD-L1 (SP263) Assay
(nr kat. 741-4905), aby zagwarantować ważność wyników analiz
obrazowych.
Ten algorytm jest przeznaczony do stosowania w diagnostyce
in vitro (IVD).
Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu 3

Znaczenie kliniczne
Rak płuc od kilku dekad jest najczęściej występującym rakiem na
świecie i pozostaje główną przyczyną zgonów wśród pacjentów
chorujących na raka na całym świecie. Szacuje się, że stanowi
on 12,9% wszystkich nowych przypadków raka i odpowiada
za blisko 1,76 miliona zgonów rocznie na całym świecie lub
około jeden na pięć zgonów wśród pacjentów chorujących na
raka. Niedrobnokomórkowy rak płuc (NSCLC) to najczęściej
występujący rodzaj raka płuc, który stanowi 85% wszystkich
przypadków raka płuc. U większości pacjentów z NSCLC
stwierdzana jest nieoperacyjna, miejscowo zaawansowana
choroba (stadium IIIB) lub choroba przerzutowa (stadium IV) —
obecnie dla żadnej z nich nie ma dostępnych opcji leczenia.
5-letni względny współczynnik przeżycia dla pacjentów z NSCLC
rozpoznanym w zaawansowanym stadium to 4,7%.
Ligand 1 programowanej śmierci (PD-L1) to białko przezbłonowe
ulegające ekspresji w aktywowanych komórkach T, które hamuje
odpowiedź immunologiczną poprzez wiązanie się do swoich
dwóch hamujących receptorów — receptora programowanej
śmierci1 (PD-1) oraz B7-1 (CD80). Wiązanie PD-L1 z PD-1
hamuje namnażanie się limfocytówT, wytwarzanie cytokin oraz
aktywność cytolityczną, co prowadzi do inaktywacji funkcjonalnej
lub wyczerpania limfocytówT. Białko PD-L1 wiąże się również
do receptora CD80 na komórkach prezentujących antygen
i aktywowanych limfocytach T, pośrednicząc w hamowaniu
odpowiedzi immunologicznych, w tym inhibicji aktywacji
komórek T i wytwarzania cytokin. Ekspresja białka PD-L1
jest obserwowana w komórkach układu immunologicznego
i komórkach TC. Zaobserwowano, żenieprawidłowa
ekspresja białka PD-L1 na komórkach TC upośledza
odpowiedź odpornościową skierowaną przeciwko komórkom
nowotworowym, awięc utrudnia możliwość ich eliminacji.
Wykazano, że wdrożenie immunoterapii, które przerywają szlak
PD-L1/PD-1, powoduje poprawę współczynników przeżycia
pacjentów z rozpoznaniem NSCLC. Rokowanie w przypadku
zastosowania leczenia zależy jednak od poziomu ekspresji białka
PD-L1. Z tego względu wymagane jest ilościowe oznaczenie
białka PD-L1 poprzez barwienie immunohistochemiczne.
Metody immunohistochemiczne mogą być stosowane w celu
wykrywania swoistych antygenów obecnych w próbkach
tkanek, stanowiąc skuteczne narzędzie używane przez
patomorfologów do stawiania rozpoznania i określania rokowania
w przypadku raka. Test VENTANA PD-L1 (SP 263) Assay zawiera
królicze przeciwciało monoklonalne przeznaczone do użytku
laboratoryjnego do półilościowej detekcji białka PD-L1 w
skrawkach FFPE prawidłowych i nowotworowych tkanek. Zaletą
preparatów histologicznych jest nienaruszona morfologia tkanek,
ułatwiająca interpretację dodatniego wyniku względem białka
PD-L1 w próbce pacjenta. Wyniki wszelkich testów
histologicznych powinny być interpretowane przez specjalistę
w dziedzinie morfologii i/lub patologii raka NSCLC. Wyniki te
powinny zostać uzupełnione o badania morfologiczne i wykonanie
odpowiednich prób kontrolnych oraz używane w połączeniu z
innymi danymi klinicznymi i laboratoryjnymi.
4 Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu

Zasady testu
Oprogramowanie uPath enterprise software wyposażone w
algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC
wykorzystuje techniki analizy obrazowej w celu uzyskania wyniku
określającego odsetek dodatnich komórek TC.
Algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC
używa wstępnie zdefiniowanych parametrów w celu oceny
obrazów tkanek wybarwionych przy użyciu testu VENTANA PD-L1
(SP263) Assay.
Etapy analizy obrazowej:
• Identyfikacja pustej przestrzeni i automatyczne wykluczenie
jej z analizy.
• Detekcja komórek na całym obrazie.
• Zaklasyfikowanie komórek do grupy komórek TC lub komórek
innego typu.
• Identyfikacja wybarwionych i niewybarwionych komórek TC.
• Obliczenie wyniku określającego odsetek dodatnich komórek
TC poprzez podzielenie liczby wybarwionych komórek TC
przez łączną liczbę komórek TC zgodnie z opisem w arkuszu
metody dla testu VENTANA PD-L1 (SP263) Assay.
Sposób identyfikacji komórek TC i obliczania wyniku przez
algorytm uPath PD-L1 (SP263) do analizy obrazów raka
NSCLC:
• Algorytm uPath PD-L1 (SP263) do analizy obrazów
raka NSCLC identyfikuje komórki TC na podstawie
koloru, intensywności wybarwienia, rozmiaru oraz cech
morfologicznych.
• Zidentyfikowane komórki TC są klasyfikowane jako komórki
wybarwione na podstawie wykrytej błony oraz wstępnie
ustawionych wartości progowych, które są zgodne z arkuszem
metody dla testu VENTANA PD-L1 (SP263) Assay.
• W celu obliczenia odsetka wybarwionych komórek TC liczba
wybarwionych komórek TC jest dzielona przez łączną liczbę
komórek TC zgodnie z opisem w arkuszu metody dla testu
VENTANA PD-L1 (SP263) Assay.
Identyfikacja pustej przestrzeni przez algorytm uPath PD-L1
(SP263) do analizy obrazów raka NSCLC:
• Algorytm automatycznie wykluczy pustą przestrzeń z analizy.
Artefakty, takie jak zabrudzenia, zarysowania lub plamy tuszu,
mogą nie zostać automatycznie wykluczone.
• Użytkownik może przejrzeć obszary określone przez
algorytm jako puste przestrzenie, używając nakładki kolorów
nierzeczywistych; patrz „Analiza obrazu po kątem białka PD-L1:
nakładka kolorów nierzeczywistych”.
Kryteria generowania wyniku określającego odsetek
dodatnich komórek TC przez algorytm uPath PD-L1 (SP263)
do analizy obrazów raka NSCLC:
• Algorytm uPath PD-L1 (SP263) do analizy obrazów raka
NSCLC będzie zgłaszał wynik z uwzględnieniem pierwszej
cyfry po przecinku, np. „4,8%”.
• Nie zaleca się zaokrąglania wyniku w górę lub w dół. Na
przykład wyniku „4,8%” nie należy zaokrąglać do 5%.
Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu 5

Algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC
jest zaprojektowany do użytku z testem VENTANA PD-L1 (SP263)
Assay. Jakość wyników testu zależy całkowicie od jakości i
dokładności zobrazowanego preparatu IHC oraz uzyskanego
obrazu, który zostaje poddany analizie.
Zanim obrazy preparatów zostaną przekazane do
oprogramowania uPath enterprise software w celu ich
analizy, patomorfolog musi zweryfikować serię barwienia
wykonywaną przy użyciu testu VENTANA PD-L1 (SP263) Assay,
przeprowadzając ręczne badanie mikroskopowe preparatów
kontrolnych PD-L1, aby ustalić, czy uzyskano oczekiwane wyniki.
Należy przestrzegać zaleceń producenta testu VENTANA PD-L1
(SP263) Assay, w tym używać wszystkich dodatnich i ujemnych
materiałów do kontroli jakości w każdej serii barwienia. Jeśli
preparaty kontrolne nie nadają się do przyjęcia na podstawie
ręcznego badania mikroskopowego, próbki tkanek należy
ponownie poddać barwieniu, aby uzyskać akceptowalne wyniki.
Patomorfolog musi przestrzegać zaleceń dotyczących interpretacji
wyniku barwienia przy użyciu testu VENTANA PD-L1 (SP263) Assay.
Patrz arkusz metody dla testu VENTANA PD-L1 (SP263) Assay (nr
kat. 1014258PL) oraz podręcznik interpretacji (nr kat. 1015317EN)
(dostępne pod adresem www.ventana.com).
Algorytm uPath PD-L1 (SP263) do analizy obrazów raka
NSCLC jest przeznaczony do użytku przez wykwalifikowanych
patomorfologów w połączeniu z wynikami badania
histologicznego, odpowiednimi informacjami klinicznymi oraz
odpowiednimi kontrolami. Nie należy z niego korzystać jak
z autonomicznego narzędzia. Proces analizy wykonywanej
za pomocą tego algorytmu musi być nadzorowany przez
kompetentną osobę.
Algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC
może generować nieprawidłowe wyniki, jeśli na zarejestrowanych
obrazach widoczne jest nietypowe wybarwienie (wybarwienie
jąder, cytoplazmy itd.).
Algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC
odrzuci wydłużone jądra komórek nowotworowych bez względu
na ogólny kształt komórki. Z tego względu zmiany nowotworowe
zawierające dużą liczbę komórek z wydłużonymi jądrami mogą
wymagać oceny ręcznej.
Ograniczenia metody
Algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC
przeszkolono, opracowano i zwalidowano na próbkach tkanek
raka NSCLC.
Nie przetestowano działania algorytmu uPath PD-L1 (SP263)
do analizy obrazów raka NSCLC w przypadku używania go z
komputerem osobistym (PC) z domu ani nie zwalidowano jego
bezpieczeństwa i skuteczności w takiej sytuacji.
Algorytm uPath PD-L1 (SP263) do analizy obrazów służy do
pomocy w identyfikacji pacjentów z rakiem NSCLC do leczenia
za pomocą terapii stosowanych w przypadku ekspresji białka
PD-L1 w przynajmniej 50% komórek TC (punkt odcięcia) zgodnie
z zatwierdzoną ChPL.
Algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC
może nieprawidłowo zidentyfikować komórki z powodu obecności
słabego wybarwienia cytoplazmatycznego i/lub błonowego,
silnego wybarwienia komórek układu immunologicznego
nakładającego się na wybarwienie komórek nowotworowych
przy znaczącym mieszanym zapaleniu, obecności komórek
TC z cytoplazmatycznym rumieńcem oraz zabarwieniu w
miejscach innych niż docelowe. Może to doprowadzić do błędnej
identyfikacji komórek TC jako komórki inne niż TC, a komórek
innych niż TC jako dodatnie komórki TC.
Chociaż konieczne jest wykluczenie makrofagów z regionu
poddawanego analizie, nie zawsze możliwe jest wykluczenie
wszystkich makrofagów. W związku z tym na wynik wygenerowany
przez algorytm uPath PD-L1 (SP263) do analizy obrazów raka
NSCLC mogą wpłynąć makrofagi obecne w analizowanym
obszarze ROI. Ma to krytyczne znaczenie, gdy wynik pacjenta jest
bliski punktu odcięcia na poziomie 50%.
Odczyn cytoplazmatyczny zwykle charakteryzuje się
rozproszonym wzorem barwienia, a w niektórych przypadkach
drobnoziarnistym wzorem barwienia. W rzadkich przypadkach
obserwowano okołojądrowe, punktowe wybarwienie o różnej
intensywności. Łączna procentowa intensywność sygnałów
z błony komórek nowotworowych jest szacowana wizualnie i
wykorzystywana do wygenerowania poziomu ekspresji białka
PD-L1. Podczas określania poziomu ekspresji białka PD-L1
nie jest uwzględniany odczyn cytoplazmatyczny komórek
nowotworowych. W celu oceny obecności tła w próbkach
testowych oraz oszacowania wyjściowej intensywności
wybarwienia stosuje się dopasowane do izotypu przeciwciało
służące jako kontrola ujemna.
6 Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu

Bezpieczeństwo danych
Złośliwe oprogramowanie lub nieuprawniony dostęp do aparatu
może doprowadzić do utraty danych lub niedostępności aparatu.
Aby uniknąć zarażenia systemu złośliwym oprogramowaniem lub
uniknąć nieuprawnionego dostępu i niewłaściwego wykorzystania
aparatu, kluczowe jest przestrzeganie następujących zaleceń:
• Nie instalować ani nie uruchamiać żadnego innego
oprogramowania w aparacie.
• Upewnić się, że inne komputery i usługi w sieci są
odpowiednio zabezpieczone i chronione przed złośliwym
oprogramowaniem i nieuprawnionym dostępem. Dotyczy to
na przykład laboratoryjnego systemu informacyjnego (LIS),
udostępniania plików archiwizacji i kopii zapasowej lub usług.
• Klienci są odpowiedzialni za bezpieczeństwo ich sieci
lokalnej, zwłaszcza w sensie jej ochrony przed złośliwym
oprogramowaniem oraz atakami. Ochrona ta może obejmować
podjęcie środków, takich jak zainstalowanie zapory sieciowej,
w celu oddzielenia urządzenia od niekontrolowanych sieci.
Ochrona ta może również obejmować podjęcie środków
umożliwiających upewnienie się, że podłączona sieć jest wolna
od złośliwych kodów.
• Ograniczać dostęp fizyczny do aparatu i całej podłączonej
infrastruktury informatycznej (komputery, kable, wyposażenie
sieciowe itd.).
• Upewnić się, że kopia zapasowa aparatu oraz pliki
archiwizacji są chronione przed nieuprawnionym dostępem
i zabezpieczone na wypadek katastrofy. Obejmuje to zdalne
przechowywanie danych, miejsca odzysku na wypadek
katastrofy i bezpieczny transfer plików kopii zapasowej.
• W razie potrzeby należy zastosować zaporę sieciową, aby
ograniczyć ruch sieciowy.
• Dysków USB można używać do wielu rodzajów tworzenia i
przywracania kopii zapasowych. Korzystanie z dysku USB
w nieprawidłowy sposób może spowodować utratę danych i
usterkę aparatu.
• Należy używać wyłącznie takich dysków USB, które zostały
przetestowane i zainstalowane przez przedstawiciela serwisu
firmy Roche.
• Można używać tylko jednego dysku USB jednocześnie.
Przed włożeniem dysku USB należy upewnić się, że do
urządzenia nie włożono już innego dysku USB.
• Przed wyjęciem dysku USB należy nacisnąć przycisk Wysuń w
systemie Windows.
• Nie należy zmieniać domyślnej konfiguracji systemu
operacyjnego (OS) dostarczonej z serwerem, ponieważ ma
to wpływ na utwardzone („hardening”) konfiguracje systemu
operacyjnego (OS).
• Aby uniknąć zarażenia oprogramowania uPath enterprise
software przez wirusa, dysku USB należy używać wyłącznie
w aparacie. Na dysku USB nie należy przechowywać żadnych
innych danych.
Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu 7

Przebieg pracy z wykorzystaniem algorytmu uPath PD-L1 do analizy obrazów
raka NSCLC
Wymagane materiały
• Oprogramowanie uPath enterprise software
• Algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC
• Preparaty tkanek raka NSCLC wybarwione przy użyciu testu VENTANA PD-L1 (SP263) Assay (z wykorzystaniem zestawu
OptiView DAB IHC Detection Kit) w aparacie BenchMark ULTRA
• Skaner preparatów VENTANA DP 200
Przebieg pracy
1. Próbka tkanki raka NSCLC znajdująca się na szkiełku podstawowym jest barwiona za pomocą testu VENTANA PD-L1 (SP263)
Assay przy użyciu aparatu BenchMark ULTRA.
2. Rejestracja obrazu (skanowanie całego preparatu) jest wykonywana za pomocą skanera preparatów VENTANA DP 200 przy
powiększeniu 20x w jednej płaszczyźnie Z.
3. Po rejestracji obrazów cyfrowych obrazy te są przesyłane z komputera skojarzonego ze skanerem preparatów VENTANA DP 200 do
systemu zarządzania obrazami (IMS) na scentralizowanym serwerze.
4. Po przesłaniu obrazów na serwer w oprogramowaniu uPath enterprise software zostanie utworzony przypadek. Przypadek może
zostać utworzony automatycznie poprzez komunikację z laboratoryjnym systemem informacyjnym (LIS) przy użyciu informacji
identyfikacyjnych (tj. typ tkanki i przeciwciało pierwszorzędowe) zawartych na etykiecie z kodem kreskowym preparatu lub
wprowadzonych ręcznie do oprogramowania uPath enterprise software (patrz dokument Oprogramowanie uPath enterprise
software — podręcznik użytkownika (nr kat. 1018943EN)).
5. Jeśli zainstalowany jest algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC (musi być zainstalowany na innym serwerze
niż oprogramowanie uPath enterprise software i system IMS), a do obrazu zarejestrowanego przy powiększeniu 20x dołączone są
informacje o odpowiednim barwniku i typie tkanki, wówczas oprogramowanie uPath enterprise software automatycznie uruchamia
analizę całego preparatu (Whole Slide Analysis, WSA).
6. W ramach analizy WSA automatycznie analizowany jest cały zeskanowany obraz.
7. Patomorfolog jest powiadamiany przez oprogramowanie uPath enterprise software o ukończeniu analizy WSA poprzez
wyświetlenie komunikatu „analysis is complete” (ukończono analizę). Patomorfolog może teraz wybrać określone obszary ROI,
które będą poddawane ocenie. Patomorfolog może wybrać jeden obszar ROI lub wiele obszarów ROI o dowolnym rozmiarze. Jeśli
wybrano wiele obszarów ROI, generowany jest sumaryczny wynik, a także indywidualny wynik dla każdego obszaru ROI.
Barwienie
• Przygotowanie i barwienie tkanek powinno odbywać się zgodnie z zaleceniami podanymi w arkuszu metody dla testu VENTANA
PD-L1 (SP263) Assay.
• Wszystkie odpowiednie kontrole powinny zostać obejrzane, a preparaty powinny zostać ponownie wybarwione, jeśli barwienie nie
spełnia wytycznych określonych w arkuszu metody dla testu VENTANA PD-L1 (SP263) Assay.
• Algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC wymaga użycia testu VENTANA PD-L1 (SP263) Assay oraz
wszelkich dodatkowych materiałów lub zasobów wymienionych w arkuszu metody dla testu VENTANA PD-L1 (SP263) Assay do
wybarwienia tkanek przed analizą. Test VENTANA PD-L1 (SP263) Assay wykrywa białko PD-L1 w skrawkach FFPE tkanki raka
NSCLC wybarwionych za pomocą zestawu OptiView DAB IHC Detection Kit w aparacie BenchMark ULTRA. Chociaż test VENTANA
PD-L1 (SP263) Assay wykrywa białko PD-L1 w skrawkach FFPE tkanki raka NSCLC wybarwionych w aparatach BenchMark
ULTRA, algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC zwalidowano, korzystając z zestawu OptiView DAB IHC
Detection Kit w aparacie BenchMark ULTRA.
Rejestracja obrazu
Do skanowania preparatów wymagany jest skaner preparatów VENTANA DP 200. Obrazy należy skanować przy powiększeniu 20x. Jeśli
duże obszary obrazu są nieostre, zalecane jest ponowne przeskanowanie preparatów. Dalsze informacje dotyczące skanowania zawiera
dokument Skaner preparatów VENTANA DP 200 — podręcznik użytkownika (nr kat. 1017149PL).
8 Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu

Ogólne informacje dotyczące poruszania się po oprogramowaniu uPath enterprise software
Możliwe jest dostosowanie oprogramowania uPath enterprise software do potrzeb indywidualnych oraz potrzeb ośrodka; istnieje m.in.
możliwość konfiguracji raportów i dostosowania interfejsu użytkownika. W tym podręczniku do algorytmu skupiono się wyłącznie na
narzędziach niezbędnych do korzystania z algorytmu uPath PD-L1 (SP263) do analizy obrazów raka NSCLC. Więcej informacji na temat
oprogramowania uPath enterprise software można znaleźć w dokumencie Oprogramowanie uPath enterprise software — podręcznik
użytkownika.
Przebieg pracy wykonywanej przez patomorfologa
Otwieranie przypadku
Dostęp do obrazów tkanki NSCLC wybarwionej za pomocą testu VENTANA PD-L1 (SP263) Assay można uzyskać, dwukrotnie klikając
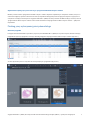
przypadek lub wybierając przypadek i naciskając zakładkę przeglądarki w oprogramowaniu uPath enterprise software (Rysunek 1).
Zostanie wyświetlony ekran ze wszystkimi obrazami powiązanymi z przypadkiem (Rysunek 2).
Rysunek 2
Rysunek 1
Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu 9

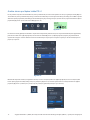
Po zeskanowaniu szkiełka podstawowego wybarwionego za pomocą testu VENTANA PD-L1 (SP263) Assay w skanerze preparatów
VENTANA DP 200 przy powiększeniu 20x obraz jest importowany do oprogramowania uPath enterprise software i kojarzony z przypadkiem.
Algorytm uPath PD-L1 (SP263) do analizy obrazów raka NSCLC automatycznie uruchomi analizę WSA. Czas trwania kroku wykonywania
wstępnych obliczeń w ramach analizy WSA zależy od specyfikacji serwera, rozmiarów obrazów i liczby obrazów w kolejce. Komunikat
„waiting to start auto-analysis” (oczekiwanie na rozpoczęcie automatycznej analizy) wskazuje, że obrazy znajdują się w kolejce, ale
jeszcze nie są analizowane, a komunikat „analyzing” (analizowanie) wskazuje, że wykonywana jest analiza WSA (Rysunek 3 i 4). Po
ukończeniu analizy obrazu w ramach analizy WSA w oprogramowaniu uPath enterprise software pod obrazem preparatu w przeglądarce
w oprogramowaniu uPath enterprise software zostanie wyświetlony komunikat „analysis successful” (analiza zakończona powodzeniem)
(Rysunek 5). Przed pomyślnym ukończeniem analizy WSA nie można wygenerować wyników dla obrazów.
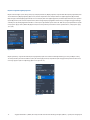
Rysowanie obszarów ROI obejmujących całą zmianę nowotworową: zaznaczanie obszaru zmiany nowotworowej
Aby wybrać obszar zmiany nowotworowej do analizy na obrazie preparatu IHC, należy użyć przycisku narzędzia Freehand (Odręcznie),
który znajduje się menu rozwijanym obszaru ROI (Rysunek 6). Na rysunku 7 przedstawiono obraz, na którym narysowano jeden obszar
ROI. Można narysować dodatkowe obszary ROI. Po zaznaczeniu każdego żądanego obszaru obszar ROI zostanie wyświetlony na panelu
Slide Panel (Panel preparatów) (Rysunek 8).
Rysunek 6 Rysunek 7
Rysunek 8
Rysunek 5
Rysunek 4
Rysunek 3
10 Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu

Rysowanie obszarów ROI obejmujących całą zmianę nowotworową: wykluczony obszar
Podczas rysowania obszarów ROI może być wymagane wykluczenie niektórych obszarów; obszary, których należy unikać lub które
należy pominąć podczas rysowania obszarów ROI, zostaną omówione, wraz z przykładami, w znajdującej się dalej części dotyczącej
charakterystyki barwienia. Aby wykluczyć określone obszary (Rysunek 10), należy użyć narzędzia wykluczenia Freehand (Odręcznie),
które znajduje się menu rozwijanym Exclusion (Wykluczanie) (Rysunek 9). Jeśli duże obszary obrazu są rozmyte lub nieostre, należy
ponownie przeskanować preparat.
Wykluczone obszary nie będą analizowane przez algorytm, a wybarwione i niewybarwione komórki TC w tym obszarze zostaną
wykluczone z całkowitego obszaru poddawanego analizie. Jeśli wykluczenie zastosowano w obszarze ROI, który został już
przeanalizowany, należy ponownie wykonać analizę obszaru ROI, aby odpowiednio zaktualizować nakładkę i wynik.
Narysowanie dużej liczby obszarów do wykluczenia — zwłaszcza jeśli są to skomplikowane obszary obrysowywane przy użyciu
narzędzia Freehand (Odręcznie) — może zająć dużo czasu i obniżyć wydajność przebiegu pracy, a tylko w minimalnym stopniu wpłynąć
na końcowy wynik. Jeśli wymagane jest narysowanie dużej liczby obszarów do wykluczenia dla przypadku, patomorfolog powinien
wykonać następujące czynności:
• Narysować wiele obszarów ROI i wykluczyć fragmenty tkanki, które w mniemaniu patomorfologa nie nadają się do oceny,
maksymalnie ograniczając korzystanie z narzędzia Exclusion (Wykluczanie).
• Ograniczyć liczbę obszarów do wykluczenia i ręcznie zastąpić wynik innym wynikiem.
Rysowanie obszarów ROI obejmujących całą zmianę nowotworową: usuwanie
Jeśli wybrany obszar ROI całej zmiany nowotworowej nie jest optymalny, można go usunąć. Wybrać obszar ROI całej zmiany nowotworowej,
klikając środek obszaru ROI na obrazie, a następnie klikając przycisk Delete (Usuń) na panelu Slide Panel (Panel preparatów) (Rysunek
11) lub na obrazie preparatu w pobliżu obszaru ROI (Rysunek 12). Zostanie wyświetlone okno z monitem o potwierdzenie. Wybrać opcję
Confirm (Potwierdź), aby usunąć wybrany obszar ROI. Wybrać opcję Cancel (Anuluj), aby zachować obszar ROI.
Rysunek 10Rysunek 9
Rysunek 11 Rysunek 12
Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu 11

Po narysowaniu wszystkich obszarów ROI całej zmiany nowotworowej i/lub obszarów wykluczenia obraz jest gotowy do analizy. Wybrać
obszar ROI całej zmiany nowotworowej, klikając środek obszaru ROI, który ma zostać przeanalizowany, lub klikając obszar ROI na panelu
Slide Panel (Panel preparatów). Dla każdego obszaru ROI wybrać przycisk Image Analysis (Analiza obrazu) na panelu Slide Panel (Panel
preparatów) (Rysunek 13) lub obok obszaru ROI (Rysunek 14).
Po ukończeniu analizy pod kątem białka PD-L1 wynik zostanie wyświetlony w dwóch miejscach na panelu Slide Panel (Panel preparatów):
pod obszarem Slide Score (Wynik preparatu) oraz obok obszarów ROI (Rysunek 15). Wynik preparatu jest oparty na podsumowaniu
wyników określających odsetek dodatnich komórek nowotworowych uzyskanych we wszystkich wybranych obszarach ROI; wynik ten
pojawi się w raporcie.
Analiza obrazu pod kątem białka PD-L1
Rysunek 15
Można także wyświetlić bardziej szczegółowe informacje na liście rozwijanej Slide Score (Wynik preparatu) oraz liście rozwijanej ROI
Details (Szczegóły obszaru ROI), klikając ikonę listy rozwijanej (Rysunek 16). Zostanie wyświetlona lista rozwijana Slide Score (Wynik
preparatu) (Rysunek 17). Kliknięcie tej samej ikony listy rozwijanej spowoduje zwinięcie tych informacji.
Rysunek 17
Rysunek 16
Rysunek 13 Rysunek 14
12 Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu

Analiza obrazu pod kątem białka PD-L1: nakładka kolorów nierzeczywistych
Na obszarze ROI zostanie wyświetlona kolorowa nakładka po naciśnięciu przycisku ROI(s) Analysis (Analiza obszarów ROI) i
przeanalizowaniu tkanki. Czerwone okręgi na poniższym obrazie (Rysunek 18) oznaczają komórki określone jako wybarwione dodatnio
względem białka PD-L1, a niebieskie okręgi oznaczają komórki określone jako ujemnie względem białka PD-L1. Podczas chwytania
obrazu (naciśnięcie lewego przycisku myszy i przesunięcie obrazu) nakładka zniknie (Rysunek 19). Po zwolnieniu przycisku myszy
nakładka pojawi się ponownie (Rysunek 18).
Rysunek 18
Rysunek 19
Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu 13

Ręczne zastąpienie wyniku preparatu
Wyniki można zastąpić ręcznie, klikając ikonę listy rozwijanej Slide Score (Wynik preparatu) na panelu Slide Panel (Panel preparatów) obok
obszaru Slide Score (Wynik preparatu) (Rysunek 16). Zostanie wyświetlona lista rozwijana Slide Score (Wynik preparatu) (Rysunek 20).
Wybranie przycisku Edit (Edytuj) (Rysunek 20) na liście rozwijanej Slide Score (Wynik preparatu) umożliwia użytkownikowi ręczne wpisanie
wyniku (Rysunek 21). Pole Comments (Komentarze) zawiera uwagi dotyczące przypadku i/lub informację o zastąpieniu wygenerowanego
automatycznie wyniku. W przypadku białka PD-L1 można ręcznie wprowadzić wyniki z zakresu 0–100%. Po ręcznym wprowadzeniu wyniku
należy wybrać opcję Confirm (Potwierdź) (Rysunek 22). Zostanie wyświetlony komunikat z monitem o potwierdzenie; należy wybrać opcję
„Yes” (Tak).
Wynik wyświetlony na panelu Slide Panel (Panel preparatów) będzie teraz odzwierciedlał wprowadzony ręcznie wynik. Wynik analizy
obrazu wyświetlany obok obszaru ROI nie będzie już widoczny (Rysunek 23). Użytkownik będzie mógł ponownie przeanalizować obraz,
naciskając przycisk wykresu słupkowego (Rysunek 13, Rysunek 14).
Rysunek 21 Rysunek 22
Rysunek 20
Rysunek 23
14 Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu

Należy zapoznać się z arkuszem metody dla testu VENTANA PD-L1 (SP263) Assay i podręcznikiem interpretacji.
Ocena pod kątem białka PD-L1
Komórki nowotworowe NSCLC wyznakowane za pomocą testu VENTANA PD-L1 (SP263) Assay są oceniane pod kątem odsetka
dodatnich komórek TC z barwieniem błonowym o dowolnej intensywności. Barwienie immunohistochemiczne preparatów raka NSCLC
powoduje wybarwienie błon komórkowych i/lub cytoplazmy i może być homogeniczne lub heterogeniczne w obrębie całej zmiany
nowotworowej. W wyniku określającym odsetek dodatnich komórek TC nie jest uwzględniany odczyn cytoplazmatyczny komórek
nowotworowych. Wzór wybarwienia błony może mieć postać nieciągłą, obwodową lub podstawno-boczną. W celu oceny obecności tła
w próbkach testowych stosuje się dopasowane do izotypu przeciwciało służące jako kontrola ujemna.
Ocena wybarwienia za pomocą algorytmu PD-L1
Patomorfolog korzystający z algorytmu uPath PD-L1 (SP263) do analizy obrazów raka NSCLC powinien być zaznajomiony z metodą
ręcznej oceny wyników testu VENTANA PD-L1 (SP263) Assay. Patomorfolog powinien użyć narzędzia Freehand (Odręcznie) do
obrysowania całego obszaru zmiany nowotworowej. Przed użyciem algorytmu uPath PD-L1 (SP263) do analizy obrazów raka NSCLC
patomorfolog powinien odnieść się do powiązanych preparatów barwionych metodą H&E oraz do ujemnych preparatów kontrolnych.
Wybierając obszary do analizy, należy wziąć pod uwagę ograniczenia metody opisane w częściach „Ograniczenia metody” i „Obszary,
których należy unikać”. Jeśli patomorfolog nie zgadza się z wynikiem podanym przez algorytm uPath PD-L1 (SP263) do analizy obrazów
raka NSCLC, powinien ręcznie zastąpić wynik.
Przypadki, których nie należy poddawać ocenie, to m. in. przypadki z niewystarczającą liczbą żywotnych komórek nowotworowych,
niedopuszczalną morfologią i tłem wprowadzającym zakłócenia. Przypadki raka NSCLC z wystarczającą liczbą żywotnych komórek TC
(według oceny patomorfologa) i bez tła wprowadzającego zakłócenia na preparatach IHC PD-L1 są dopuszczalne do oceny.
Charakterystyka barwienia
Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu 15

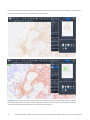
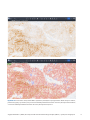
Rysunek 24: Zeskanowane obrazy tkanki NSCLC wybarwionej metodą IHC w oprogramowaniu uPath enterprise software;
przed analizą (wyżej) i po analizie (niżej). Czerwona nakładka przedstawia komórki TC określone jako wybarwione dodatnio,
a niebieska nakładka przedstawia komórki TC określone jako wybarwione ujemnie.
Na kolejnych rysunkach (24–25) przedstawiono obrazy różnych wzorów barwienia i poziomów ekspresji białka PD-L1. Ekspresja białka
PD-L1 w komórkach TC jest zgłaszana jako wartość procentowa (0–100).
16 Algorytm uPath PD-L1 (SP263) do analizy obrazów niedrobnokomórkowego raka płuc (NSCLC) — podręcznik do algorytmu
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
Strona się ładuje...
-
 1
1
-
 2
2
-
 3
3
-
 4
4
-
 5
5
-
 6
6
-
 7
7
-
 8
8
-
 9
9
-
 10
10
-
 11
11
-
 12
12
-
 13
13
-
 14
14
-
 15
15
-
 16
16
-
 17
17
-
 18
18
-
 19
19
-
 20
20
-
 21
21
-
 22
22
-
 23
23
-
 24
24
-
 25
25
-
 26
26
-
 27
27
-
 28
28
-
 29
29
-
 30
30
-
 31
31
-
 32
32
-
 33
33
-
 34
34
-
 35
35
-
 36
36
-
 37
37
-
 38
38
Roche uPath PD-L1 image analysis for NSCLC IVD Algorithm Instrukcja obsługi
- Typ
- Instrukcja obsługi
Powiązane artykuły
-
Roche BenchMark XT/LT Interpretation Guide
-
Roche uPath IVD Instrukcja obsługi
-
Roche BenchMark XT/LT Instrukcja obsługi
-
Roche BenchMark ULTRA Instrukcja obsługi
-
Roche BenchMark GX Instrukcja obsługi
-
Roche Ventana System Software (VSS) Instrukcja obsługi
-
Roche cobas s 201 system Instrukcja obsługi
-
Roche cobas s 201 system Instrukcja obsługi
-
Roche Virtuoso Instrukcja obsługi
-
Roche Virtuoso Instrukcja obsługi






































